今年公开的论文中同时出现了两个名为PEPSEEK的技术工具——PEPSeek发表于Molecular Cellular Proteomics(链接:https://www.sciencedirect.com/science/article/pii/S1535947625000350);PepSeek来自生物预印本平台bioRxiv(链接:https://www.biorxiv.org/content/10.1101/2025.04.29.641945v1)。
PEPSeek,全称Pathogen EPitope Seeker,专注于病原体感染模型中MHC-I免疫肽组学的表位识别,而PepSeek则致力于构建一个通用肽段发现框架。这种差异本质上反映了肽段研究领域的两个核心挑战:一是如何从复杂的质谱数据中精准识别低丰度肽段,二是如何在有限数据条件下实现肽段功能的准确预测与设计。

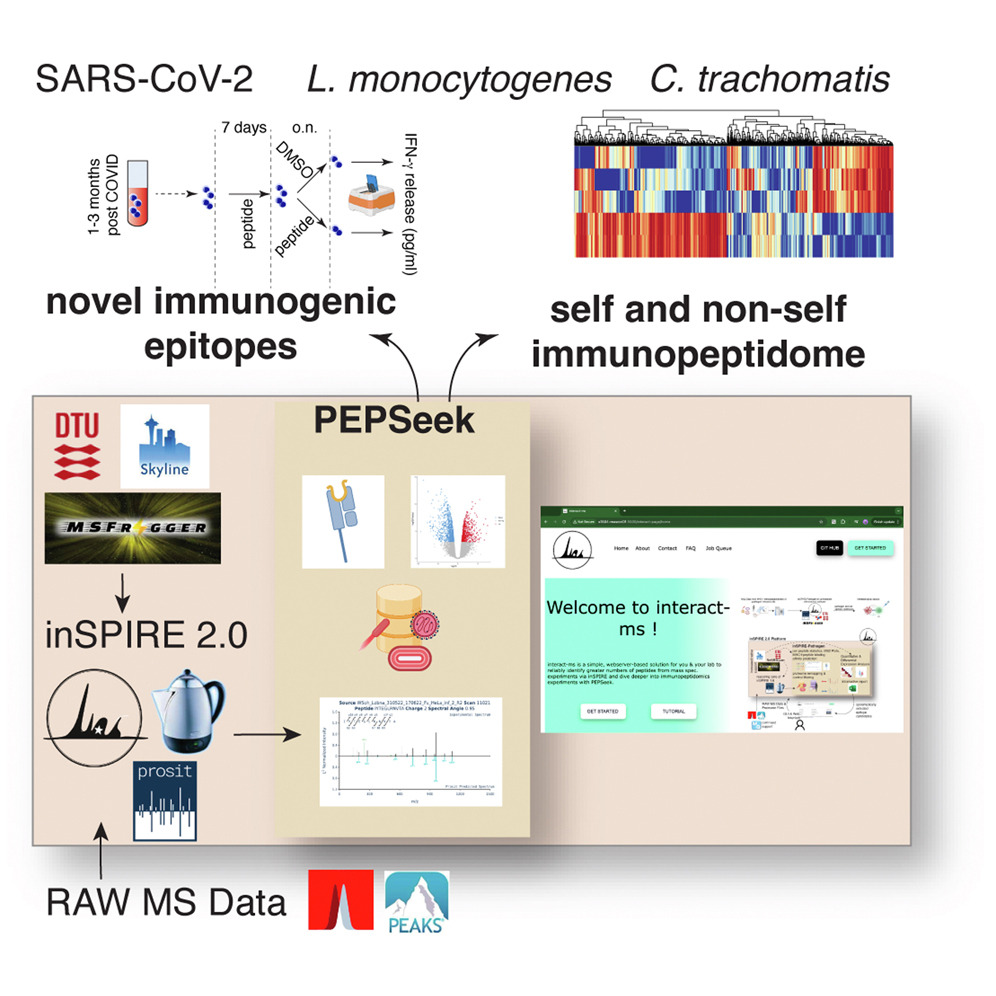
PEPSeek由德国马普所的Juliane Liepe 等人牵头研发,其工作建立在升级的inSPIRE工作流程之上,后者集成了Prosit谱图预测和Percolator重打分算法。PEPSeek的创新之处在于将专业分析流程封装成交互式网页服务器,使不具备计算背景的研究者也能轻松进行病原体表位识别、并直接输出表位候选列表。值得注意的是,PEPSeek在肽段识别中引入了后验错误概率而非传统的q值cutoff作为阈值,这一改进显著降低了假阳性率;但这并不是首创。
研究团队将PEPSeek应用于三种重要病原体感染模型:SARS-CoV-2、单核细胞增生李斯特菌和沙眼衣原体。通过分析感染和未感染的人类及小鼠细胞系的MHC-I免疫肽组学数据,与标准分析方法相比,PEPSeek识别出的病原体衍生表位候选物数量增加了57%,抗原数量增加了38%。在SARS-CoV-2感染细胞中,研究人员发现了24个新候选表位。研究团队合成了8个表位候选肽段并在COVID-19康复者的外周血单核细胞中进行测试。结果显示其中7个肽段能够触发显著的IFN-γ分泌反应,证实了这些表位的免疫原性并体现了PEPSeek的数据挖掘表现。
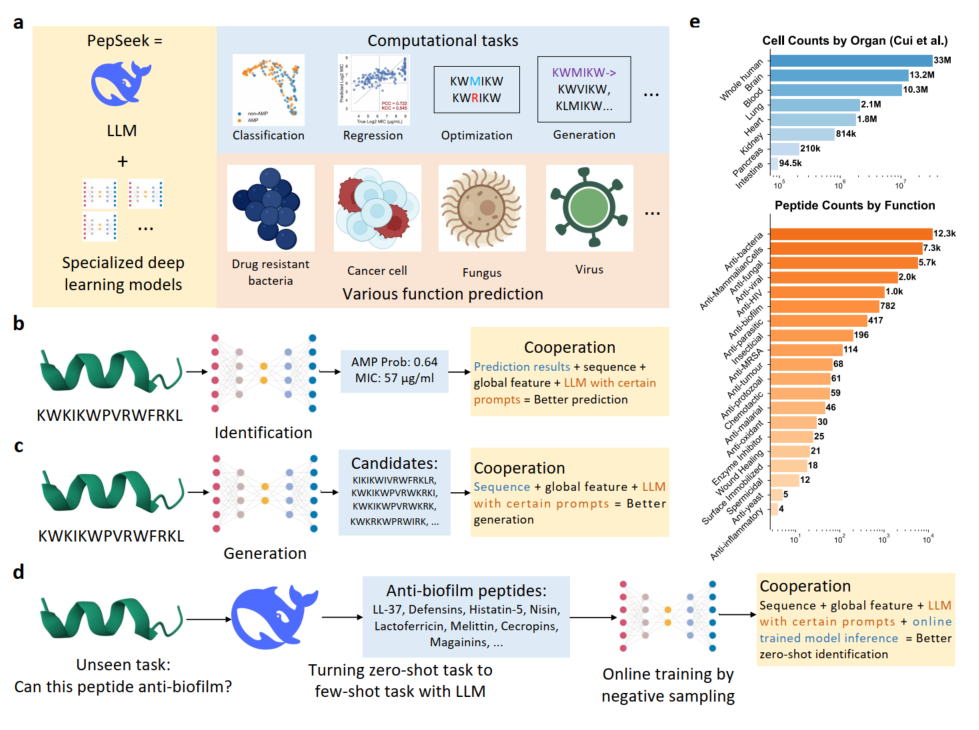
PepSeek技术由山东大学的李延青等人牵头,基于大语言模型和专门深度学习模型的协同,旨在解决肽段数据稀缺和”域外”泛化问题。前者具有强大的推理能、后者具有高精度预测能力,因此该框架理论上对于肽段识别、生成和优化等多重任务是通用的。
在肽段识别任务中,专门深度学习模型的预测结果会由大型语言模型进一步精炼,从而提升准确率。在肽段生成方面,系统采用先验引导的强化学习产生候选序列,然后由LLM进行活性优化和毒性降低。具体到肽段分类和回归任务,PepSeek采用了LLM引导的模型集成策略,根据验证性能选择最优模型子集。研究结果显示,在抗菌肽识别任务中,这种协同方法将域外数据的AUC从0.71提升至0.9以上。在实践应用中,研究团队使用该技术优化了一个源肽段QLX-227-1,生成了十大候选肽(R1-R10)。其中R2肽段在实验中表现出优异的广谱抗菌活性,对多种耐药菌的最小抑制浓度几何均值低至7.1μg/mL,优于所有处于临床试验阶段的抗菌肽。同时,该肽段具有低溶血毒性特征,治疗窗口宽广,且不易诱导耐药性产生。
两项技术虽然名称相似,但在目标定位、数据基础和方法论上存在本质差异。PEPSeek专注于质谱数据分析,强调对实验数据的精准解读;而PepSeek立足于序列信息挖掘,注重对肽段功能的预测与设计。前者需要高质量的质谱数据作为输入,后者则依赖于序列特征和理化性质。这种差异使得它们分别适用于不同的研究场景和科学问题。
然而,这种差异性也为二者的技术融合提供了想象空间:研究人员首先使用PEPSeek处理感染细胞的质谱数据,识别出候选表位;然后将这些肽段序列输入PepSeek的大型语言模型模块,进行零样本功能预测,评估其交叉反应性或毒性特征,从而优先选择最具潜力的候选物进行后续实验。在个性化医疗和疫苗开发场景中,这种联用策略或许能够降低数据检测上的假阳性、并提高生物应用上的成功率。例如:研究人员可以利用PEPSeek的检测能力监测感染过程中的病原丰度变化,同时借助PepSeek的生成式模型优化肽段设计(如对特定基因型的多肽呈递偏好),增强免疫原性或降低脱靶效应。
Content Refined by AI tools 内容经AI工具修改,审慎阅读
Figure Generated by AI tools 图像由AI工具生成